

 HOME | ÍNDICE POR TÍTULO | NORMAS PUBLICACIÓN
HOME | ÍNDICE POR TÍTULO | NORMAS PUBLICACIÓN Espacios. Vol. 37 (Nº 34) Año 2016. Pág. 7
Sergio Silva RIBEIRO 1; Maria Salete Marcon Gomes VAZ 2; Luciane HENNEBERG 3; David de Souza JACCOUD FILHO 4; Marcio Hosoya NAME 5; Rosane FALATE 6
Recibido: 22/06/16 • Aprobado: 01/082016
RESUMO: O Brasil é um dos maiores produtores mundial de milho e sua classificação comercial é regulamentada pela quantidade de grão ardidos. O objetivo deste trabalho é identificar grãos ardidos através da combinação de técnicas de mineração de dados e de processamento digital de imagens, analisando histogramas das imagens com diferentes algoritmos de mineração. Além disso, foram identificados quais algoritmos apresentavam melhores resultados para a identificação de grãos ardidos. Foram utilizados 126 grãos de milho, sendo 50% de grãos sadios e 50% de grãos ardidos. Foi utilizado um escâner para obtenção das imagens individuais de grãos, que foram processadas para extração dos histogramas e geração da base de dados. Com o Software Weka, a base foi submetida a diferentes algoritmos de mineração de dados, com geração de uma solução, e posterior testes de classificação. Os algoritmos que apresentaram melhores resultados, com taxas de acertos acima de 90%, foram NaiveBayes e NaiveBayesUpdateables. Como resultado deste artigo, constata-se que os histogramas das imagens, em conjunto com algoritmos de mineração de dados, podem apoiar na identificação de grãos ardidos de milho. |
ABSTRACT: Brazil is one of the largest global producers of corn and its commercial classification is regulated by the amount of rot grain. The objective of this study was to identify rot grain by combining techniques of data mining and digital image processing, analyzing histograms of images with different mining algorithms. In addition, we identified what algorithms showed better results for the identification of rot grain. 126 maize grains were used, 50% of healthy grains and 50% rot grain. A scanner for obtaining images of individual grains, processed to extract histograms and generating the database has been used. With the Weka software, the base was subjected to different data mining algorithms, generating a solution, and subsequent classification tests. The algorithms performed better, with hit rates above 90%, were NaiveBayes and NaiveBayesUpdateables. As a result of this work, it is verified that histograms of the images, together with data mining algorithms, can assist in the identification of rot corn grain. |
O Brasil é um dos maiores produtores de milho no mundo, com produção estimada em 93,6 milhões de toneladas para 2022/23 (Santos et. al. 2002; Brasil, 2016; Ribeiro, 2014). Até 2050 a produção de milho no mundo deverá dobrar e, por volta de 2025, deverá se tornar a cultura com maior produção nos países em desenvolvimento (Cgiar, 2016). Segundo Cruz et. al. (2011), a importância econômica do milho se dá pela grande variedade de aplicação, desde a alimentação animal até a indústria de alta tecnologia. O uso de grãos de milho, na alimentação animal, representa o maior consumo no mundo. No Brasil, o consumo animal varia de 70 a 90%, dependendo da região geográfica e, embora o uso na alimentação humana seja menor que 30%, ainda assim é um cereal de grande valor, sobretudo, para a população de baixa renda.
A preocupação dos produtores de milho está nas perdas de produtividade, qualidade, palatabilidade e valor nutritivo, devido ao ataque de patógenos, intensificados a partir da década de 90. Os grãos ardidos de milho, atacados por patógenos, possuem pelo menos um quarto da superfície com descoloração, cujo matiz pode variar do marrom claro ao roxo ou, do vermelho claro ao vermelho intenso. A causa da descoloração está relacionada à podridão das espigas, acometidas por fungos (Pinto, 2005).
Os fungos mais frequentemente encontrados nos grãos ardidos são Fusarium verticilioides, Diplodia maydis (Stenocarpella maydis) e Diplodia macrospora (Stenocarpella macrospora), atuando de forma deletéria na qualidade dos grãos (Costa et. al. 2003; Mendes et. al. 2012). A partir de setembro de 2013, entraram em vigência as instruções normativas IN MAPA nº 60/2011 e 18/2012, que estabelecem os limites máximos de tolerância de 1,0% para os grãos ardidos, do total de 15,0 % dos grãos avariados (Conab, 2013).
O processamento digital de imagens (PDI) é utilizado em diversas áreas, como, por exemplo, na computação (Valle et. al., 2013; Name et. al., 2014) e agronomia (Name, 2013; Name et. al., 2016). O PDI auxilia na tomada de decisões por meio de informações visuais, obtidas na forma de imagens digitais. O menor elemento de uma imagem é denominado de pixel. Um histograma de uma imagem, convertida para a escala de cinza, é a composição do percentual ou quantidade total de pixels para cada valor desta escala (Marques Filho, Vieira Neto, 1999; Gonzalez, Woods, 2010).
A mineração de dados corresponde ao processo de busca de padrões consistentes, que se repetem por meio de regras ou sequências temporais em um grande volume de dados (Jiawei, Kamber, 2011; Pacheco et. al. 2013). E, são utilizadas técnicas de estatística, inteligência artificial, recuperação de informação, algoritmos de aprendizagem, rede neurais, rede bayesiana, entre outras (Dziekaniak, 2010). O software de mineração de dados Weka (Hall et. al. 2009) possui uma interface amigável e capacidade de processamento rápido. A vantagem é oferecer diversos algoritmos de mineração de dados em seu ambiente, além de apresentar possibilidade de criação de novos algoritmos. Os principais algoritmos disponíveis são de regressão, classificação, mineração de regras de associação, e seleção de atributos (Sato et. al. 2013).
Embora que, a solução do problema de grãos ardidos seja importante para a agricultura, existem poucos trabalhos de uso de métodos computacionais para este fim. O objetivo deste trabalho é aplicar algoritmos e/ou métodos de mineração de dados para detecção de grãos ardidos de milho.
A metodologia deste trabalho envolve a aplicação dos algoritmos de mineração de dados, em uma base de dados de histogramas de imagens de grãos de milho, para identificação de grãos ardidos; com posterior análise comparativa dos algoritmos, com exame do grau de acerto e do tempo de processamento de cada um deles.
Os experimentos foram realizados no Laboratório de Sistemas Digitais e no Laboratório de Fitopatologia Aplicada, pertencentes à UEPG – Universidade Estadual de Ponta Grossa. O Laboratório de Fitopatologia Aplicada forneceu 126 amostras de grãos de milho, sendo 63 sadios, Figura 1(a), e 63 correspondentes aos grãos ardidos, Figura 1(b).
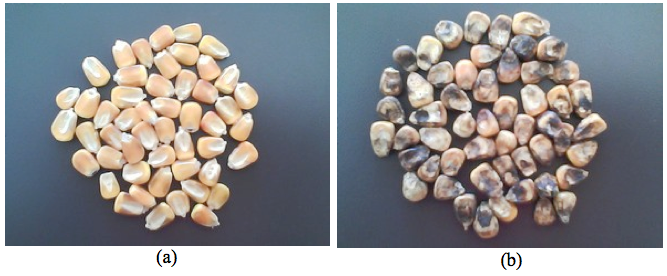
Figura 1 – (a) Grãos sadios e (b) ardidos utilizados no experimento.
Para a aquisição das imagens foi utilizado um escâner SAMSUMG, Modelo SCX-4600 (Figura 2), preparado com marcadores para posicionamento dos grãos e foi coberto o escâner com um tecido (feltro) preto Figura 2(b), para reduzir/eliminar a entrada da luminosidade, podendo influenciar a aquisição da imagem
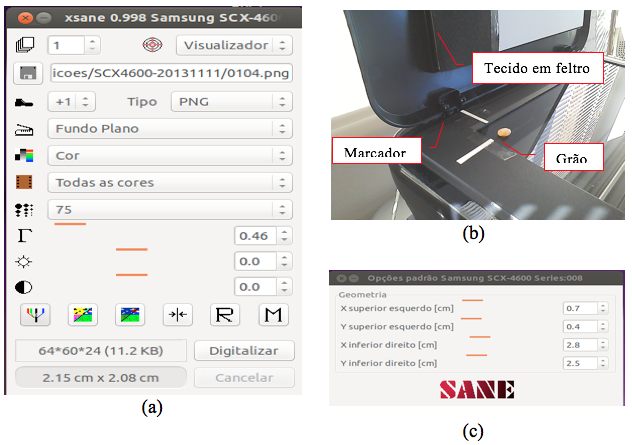
Figura 2 - Scanner SCX-4600 preparado para aquisição de imagens, parametrizado no Xsane:
(a) Configurações de imagem, (b) Adaptações do escâner para as aquisições e (c) Localizações da área de digitalização.
As aplicações foram executadas em um Notebook ACER Aspire, Modelo 5733-6663, com Processador Intel Core i3 e 4GB de memória, com Sistema Operacional Linux UBUNTU Versão 13.10.
A aquisição das imagens foi feita com o auxilio do Software XSane Imaging Scanner Program, Versão 0.998, com a configuração da imagem (Figura 2(a)) como segue. O tipo de arquivo png, com fundo plano, possibilitando todas as cores, com resolução de 600 dpi, com dimensões 504x536x24, e localização da área de digitalização no escâner, Figura 2(c), onde x superior esquerdo com 0,3 cm, y superior esquerdo com 0,2 cm, x inferior direito com 2,5 cm, e y inferior direito com 2,5 cm.
O tempo médio para aquisição de cada grupo de grãos foi de 25 minutos. Antes da utilização do Software XSane, para a digitalização da imagem, foi realizada a verificação do posicionamento do grão na bandeja do escâner. Isso foi necessário devido à anatomia dos grãos de milho, que frequentemente escorregavam na bandeja, saindo da posição inicial.
As imagens foram processadas por um programa próprio, desenvolvido na Linguagem de Programação Java e a API - Application Program Interface – Interface do Programa de Aplicação, JAI - Java Advanced Imaging – Manipulação Avançada de Imagens com Java,possibilitando a representação, o processamento e a visualização de imagens (Santos, 2004).
Primeiramente, o programa realizou a leitura do arquivo DADOS, em formato texto, contendo os dados referentes às imagens, com os dados categorizados em SADIO e ARDIDO. Após a identificação das imagens, de acordo com sua categoria, o programa convertia as imagens em tons de cinza, pela média entre os canais R (Red/Vermelho), G (Green/Verde) e B (Blue/Azul). Com base nos valores convertidos para a escala de cinza, foi gerado o histograma de cada imagem. Em seguida, esses valores foram separados, individualmente, por imagem, para a geração do arquivo .arff, a ser lido pela Ferramenta Weka. Por fim, foram utilizados os arquivos .arff como entrada para o treinamento e teste na Ferramenta Weka de mineração de dados.
Para realizar a mineração de dados, foi utilizada a Versão 3.6.6 do Weka, onde foram avaliados os 59 métodos de classificação existentes na ferramenta, separados em 7 grupos, como segue: Bayes (8 métodos), Functions (7 métodos), Lazy (4 métodos), Meta (15 métodos), Misc (2 métodos), Rules (9 métodos), e Trees (14 métodos).
Os algoritmos de aprendizagem de máquina exercitados, a partir do conjunto de dados e sua aplicação, são obtidos os parâmetros Correctly Classified, correspondendo ao percentual de itens classificados de forma correta; Relative absolute error, correspondendo ao erro relativo absoluto; e Time Taken(s), correspondendo ao custo de processamento de cada algoritmo em segundos (Costa et. al. 2010). Outro parâmetro é o Índice Kappa, que é uma medida de associação utilizada para descrever e testar o grau de confiabilidade e precisão de uma classe (Kotz, Johnson, 1983).
O Arquivo .arff gerado pelo algoritmo foi carregado no Weka, por meio da opção “explorer”. Os dados foram lidos pela ferramenta, que reconheceu 126 instâncias (grãos), com 257 atributos, 1 referente à Classe Sadio ou Ardido e, 256 correspondentes aos valores de cinza gerados pelo histograma de cada registro/imagem.
Após o armazenamento do arquivo de treino, foi selecionado o Método Holdout, para treinamento e teste, onde 2/3 dos dados da base são destinados para treinamento e 1/3 dos dados são para validação do modelo (Olson, Delen, 2008). Finalmente, cada um dos 71 métodos foi executado.
Posteriormente a execução dos 71 métodos, os dados dos algoritmos com taxa de acerto superior a 80% foram sintetizados na Tabela 1. Os métodos do Grupo Bayes apresentaram taxa de acerto superior a 90%, com 6 métodos. O Grupo Meta com 3, e Misc e Trees com 1.
Tabela 1: Comparativo do resultado do Método Bayes.
Grupo |
Algoritmo |
Taxa de Acerto (%) |
Erro relativo Absoluto (%) |
Kappa |
Tempo de Processamento (s) |
Bayes |
NaiveBayes |
93 |
14 |
0,860 |
0,08 |
NaiveBayesUpdateable |
93 |
14 |
0,860 |
0,01 |
|
BayesNet |
91 |
19 |
0,814 |
0,07 |
|
ComplementNaiveBayes |
91 |
19 |
0,814 |
0,03 |
|
NaiveBayesMultinomial |
91 |
19 |
0,814 |
0,02 |
|
NaiveBayesMultinomialUpdateable |
91 |
19 |
0,814 |
0,02 |
|
BayesianLogisticRegression |
88 |
23 |
0,767 |
0,05 |
|
Meta
|
Dagging |
91 |
43 |
0,814 |
0,19 |
MultiBoostAB |
91 |
25 |
0,813 |
0,11 |
|
RandomSubSpace |
91 |
53 |
0,813 |
0,12 |
|
AdaBoostM1 |
88 |
31 |
0,766 |
0,10 |
|
RotationForest |
88 |
37 |
0,767 |
2,27 |
|
AttributeSelectedClassifier |
86 |
38 |
0,720 |
0,34 |
|
ClassificationViaRegression |
86 |
42 |
0,721 |
0,35 |
|
FilteredClassifier |
86 |
37 |
0,720 |
0,04 |
|
RandomCommittee |
84 |
43 |
0,674 |
0,09 |
|
Bagging |
81 |
52 |
0,629 |
0,36 |
|
Misc |
HyperPipes |
91 |
97 |
0,814 |
0,01 |
VFI |
86 |
71 |
0,721 |
0,04 |
|
Trees
|
LMT |
91 |
26 |
0,813 |
1,60 |
FT |
88 |
24 |
0,766 |
0,30 |
|
RandomTree |
88 |
23 |
0,767 |
0,00 |
|
NBTree |
86 |
32 |
0,721 |
16,25 |
|
REPTree |
86 |
40 |
0,720 |
0,14 |
|
ADTree |
84 |
47 |
0,674 |
0,21 |
Os métodos com melhores resultados foram NaiveBayes e NaiveBayesUpdateables, onde obtiveram taxa de acerto de 93%. Entretanto, quando foi observado o tempo de processamento, foi verificado que o Naive/BayesUpdateables teve desempenho 8 (oito) vezes superior ao NaiveBayes.
Considerando um cenário, com grande volume de dados, onde tempo de processamento é crítico, devem ser ponderados os parâmetros taxa de acerto e tempo de processamento.
Towell et. al. (1990) propuseram uma abordagem hibrida conhecida como KBANN - Knowledge Based Neural Network, baseada em redes neurais e regras simbólicas, a qual compara rede neural do tipo perceptron multicamadas, árvore de decisão induzida pelos Algoritmos ID3 e k-NN, e a Técnica O'Neill. Em seus resultados, as redes neurais apresentaram os melhores resultados com 96,22% de acurácia para KBANN e 92,45% para perceptron multicamadas.
Tavares et. al. (2007) compararam os métodos de aprendizado de máquina, aplicados na detecção de regiões promotoras de DNA da bactéria Escherichia coli. Os métodos Bayesianos e HMM, de uma forma geral, apresentaram os melhores resultados, próximos aos de Towell et. al. (1990), considerando redes neurais.
Sato et. al. (2013) compararam algoritmos de árvore de decisão para classificação do uso e cobertura de terra. Obtiveram o melhor resultado com o SimpleCart, com melhor classificação dos pixels e melhor resultado no Índice Kappa. O algoritmo SimpleCart apresentou estrutura compatível com o Sistema ENVI - Environment for Visualizing Images.
Neste trabalho foram executados 71 algoritmos, o que possibilitou uma análise comparativa dos vários métodos computacionais de mineração de dados. Os métodos de aprendizado de máquina apresentaram resultados próximos aos de Towell et. al. (1990) e Tavares et. al. (2007) e diferentes dos resultados de Sato et. al. (2013). Os algoritmos de árvore de decisão não apresentaram bons resultados, principalmente o SimpleCart. Todavia o LMT, na análise realizada neste trabalho obteve 91% de acurácia.
Este trabalho foi parcialmente financiado pela Coordenação de Aperfeiçoamento de Pessoal de Nível Superior (CAPES), pela Fundação Araucária de Apoio ao Desenvolvimento Científico e Tecnológico do Estado do Paraná, e pelo Conselho Nacional de Desenvolvimento Científico e Tecnológico (CNPq).
BRASIL. Ministério da Agricultura. Milho. [Citado em 04 de junho de 2016]. Disponível na World Wide Web: http://www.agricultura.gov.br/vegetal/culturas/milho.
CGIAR. Research Program MAIZE 2014 Annual Report. Mexico, D.F.: CIMMYT. [Citado em 04 de junho de 2016]. Disponível na World Wide Web: http://library.cgiar.org/bitstream/handle/10947/2857/Annual_progress_report_2014_CGIAR_Research_Program_on_Maize_(MAIZE).pdf.
CONAB. Novo padrão de classificação de milho. Comunicado n.º 194/2013. [Citado em 14 de maio de 2016]. Setembro de 2013. Disponível na World Wide Web: http://www.conab.gov.br/OlalaCMS/uploads/arquivos/13_09_02_17_38_42_comu_194.pdf.
COSTA, R. S.; Môro, F. V.; Môro, J. R.; Da Silva, H. P.; Panizzi, R. D. C. (2003); “Relação entre características morfológicas da cariopse e fusariose em milho”, Pesquisa Agropecuária Brasileira, Brasília, 38 (1), 27-33.
COSTA, F. D. O.; Motta, L. C. S.; Nogueira, J. L. T. (2010); “Uma abordagem baseada em Redes Neurais Artificiais para o auxílio ao diagnóstico de doenças meningocócicas”, Revista Brasileira de Computação Aplicada, 2 (1), 79-88.
CRUZ, J. C.; Pereira Filho, I. A.; Pimentel, M. A. G.; Coelho, A. M.; Karam, D.; Cruz, I.; Garcia, J. C.; Moreira, J. A. A.; Oliveira, M. F. de; Gontijo Neto, M. M.; Albuquerque, P. E. P. de; Viana, P. A.; Mendes, S. M.; Costa, R. V. da; Alvarenga, R. C.; Matrangolo, W. J. R. (2011), “Produção de milho na agricultura familiar”, Sete Lagoas: Embrapa Milho e Sorgo, 159, 42p.
DZIEKANIAK, G. (2010); “Tecnologias de descoberta de conhecimento na gestão do conhecimento: contextualizações com a sociedade do conhecimento”, Datagrama Zero, 11 (1).
GONZALEZ, R. C.; Woods, R. E. (2010); Processamento Digital de Imagens. 3 ed.,
São Paulo: Pearson Prentice Hall, 976 p.
HALL, M.; Frank, E.; Holmes, G.; Pfahringer, B. Reutemann, P.; Witten, I. H. (2009); “The Weka Data Mining Software: An Update”, SIGKDD Explorations, 11 (1).
JIAWEI, H.; Kamber, M. (2011); Data Mining: Concepts and Techniques. 3 ed., Morgan Kaufmann, 744 p.
KOTZ, S.; Johnson N. L. (1983); Encyclopedia of statistical sciences. New York: John Wiley & Sons, 672 p.
MARQUES FILHO, O.; Vieira Neto, H. (1999); Processamento Digital de Imagens. Rio de Janeiro: Brasport, 307 p.
MENDES, M. C.; Von Pinho, R. G.; Von Pinho, E. V. R.; Faria, M. V. (2012); “Comportamento de híbridos de milho inoculados com os fungos causadores do complexo grãos ardidos e associação com parâmetros químicos e bioquímicos”, Ambiência, Guarapuava, 8 (2), 275-292.
NAME, M. H. (2013); Método computacional para avaliação do crescimento radicular da cultura da soja, 62 p. Dissertação (Mestrado em Computação Aplicada) – Setor de Ciências Agrárias e de Tecnologia, Universidade Estadual de Ponta Grossa, Ponta Grossa.
NAME, M H.; Ribeiro, S. S.; Maruyama, T. M.; Valle, H. P.; Falate, R.; Vaz, M. S. M. G. (2014); “Metadata Extraction for Calculating Object Perimeter in Images”, Revista IEEE América Latina, 12 (8), 1566-1571.
NAME, M H.; Martins Júnior, H. L.; Maruyama, T. M.; Falate R. (2016); “Desenvolvimento e comparação entre softwares destinados à avaliação do comprimento radicular”, Espacios, 37 (4), 22.
OLSON, D.; Delen, O. D. (2008); Advanced Data Mining Techiniques. Springer, 180 p.
PACHECO, D. A. J.; Machado L.; Jung, C. F.; Ten Caten, C. S. (2013); “Investigando o uso da mineração de dados nos processos de gestão da qualidade total: um estudo de caso na indústria”, Espacios, 34 (6), 15.
PINTO, N. F. J. A. (2005); “Grãos ardidos em milho”, Sete Lagoas: Embrapa Milho e Sorgo, 66, 1p.
RIBEIRO, S. S. (2014); “Cultura do Milho no Brasil”, Revista Científica Semana Acadêmica, 49 (1), 1-13.
SANTOS, P. G.; Juliatti, F. C.; Buiatti, A. L.; Hamawaki, O. T. (2002); “Avaliação do desempenho agronômico de híbridos de milho em Uberlândia, MG, Pesquisa Agropecuária Brasileira, Brasília, 37 (5), 597-602.
SANTOS, R. (2004); “Java Advanced Imaging API: A Tutorial”, Revista de Informática Teórica e Aplicada, Rio Grande do Sul, 11 (1), 93-123.
SATO, L. Y.; Shimabukuro, Y. E.; Kuplich, T. M.; Gomes, V. C. F. (2013); “Análise comparativa de algoritmos de árvore de decisão do sistema WEKA para classificação do uso e cobertura da terra”, In: XVI Simpósio Brasileiro de Sensoriamento Remoto - SBSR, Foz do Iguaçu, 2353-2360.
TAVARES, L. G.; Lopes, H. S.; Lima, C. R. E. (2007); “Estudo comparativo de métodos de aprendizado de máquina na detecção de regiões promotoras de genes de escherichia coli”, In: I Simpósio Brasileiro de Inteligência Computacional, Florianópolis.
TOWELL, G. G.; Shavlik, J. W.; Noodewier, M. O. (1990); “Refinement of approximate domain theories by knowledge-based artificial neural networks”, In: Proceedings of the 8th National Conference on Artificial Intelligence (AAAI-90), 2, 861–866.
VALLE, H. P.; Maruyama, T. M.; Name, M. H.; Falate, R. (2013); “Development of Image Segmentation Method Through Contour Algorithms”, Iberoamerican Journal of Applied Computing, 3 (3), 1-12.
1. (UEPG), professor@sergioribeiro.com.br
2. (UEPG), salete@uepg.br
3. (UEPG), lucihenne@gmail.com
4. (UEPG), dj1002@uepg.br
5. (IFC), name@ifc-camboriu.edu.br
6. (UEPG), rfalate@gmail.com